Описание задачи
Данная задача посвящена созданию модели мембраны DPPC.
Цель.
Создание модели мембраны DPPC, а затем минимизация системы и расчет молекулярной динамики в NVT и NPT ансамблях.
Расчеты осуществляются при помощи программного пакета Gromacs.
Подготовка к работе
Для выполнения задачи необходим компьютер, работающий под OS Linux, либо родственными ОС. Расчеты молекулярной динамики рекомендуется выполнять на высокопроизводительных компьютерных кластерах.
Необходимое ПО :
1 Gromacs - осуществляет расчеты молекулярной динамики (в данной статье использована версия 4.5.4) как в обычной, так и в MPI версиях (для распределенных вычислений на кластере)
2 Gnuplot - используется для построения графиков (в статье рассмотрена версия 5)
3 LAM-MPI - драйвер, необходимый для распределенных вычислений
Для удобства работы рекомендуем использовать файловые менеджеры, например, Midnigt Commander.
Описание системы и ее подготовка.
Система представляет собой мембрану из Дипальмитоилфосфатидилхолина (Dipalmitoylphosphatidylcholine, DPPC).
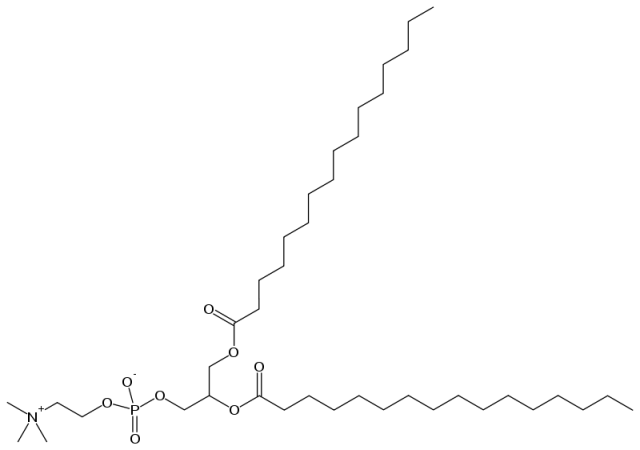 структурная формула DPPC
структурная формула DPPC
Pdb-файл с системой, состоящей из молекул DPPC и воды, Вы можете скачать с сайта D. Peter Tieleman. В данной задаче был использован файл dppc128.pdb, содержащий 128 молекул DPPC и 3655 молекул воды.
Так же скачайте следующие файлы:
- dppc.itp - файл топологии для DPPC
- lipid.itp - параметры Berger lipid
Чтобы использовать параметры lipid.itp необходимо внести некоторые изменения в уже существующий gromos53a6.ff/forcefield.itp.
Создайте в вашей рабочей директории новую папку с названием gromos53a6_lipid.ff. Скопируйте в новую папку следующие файлы из gromos53a6.ff:
- aminoacids.rtp
aminoacids.hdb
aminoacids.c.tdb
aminoacids.n.tdb
aminoacids.r2b
aminoacids.vsd
atomtypes.atp
ff_dum.itp
ffnonbonded.itp
ffbonded.itp
forcefield.itp
ions.itp
spc.itp
watermodels.dat
Затем необходимо создать forcefield.doc - файл, содержащий описание силового поля. Например, можно записать так:
GROMOS96 53A6 force field, extended to include Berger lipid parameters
Далее необходимо скопировать содержимое разделов [ atomtypes ], [ nonbond_params ] и [ pairtypes ] из lipid.itp в соответствующие разделы ffnonbonded.itp. Вы заметите, что в содержимом [ atomtypes ] из lipid.itp отсутствует колонка с номерами атомов, ее необходимо будет добавить так, чтобы скопированное содержимое lipid.itp выглядело следующим образом:
LO 8 15.9994 0.000 A 2.36400e-03 1.59000e-06 ;carbonyl O, OPLS LOM 8 15.9994 0.000 A 2.36400e-03 1.59000e-06 ;carboxyl O, OPLS LNL 7 14.0067 0.000 A 3.35300e-03 3.95100e-06 ;Nitrogen, OPLS LC 6 12.0110 0.000 A 4.88800e-03 1.35900e-05 ;Carbonyl C, OPLS LH1 6 13.0190 0.000 A 4.03100e-03 1.21400e-05 ;CH1, OPLS LH2 6 14.0270 0.000 A 7.00200e-03 2.48300e-05 ;CH2, OPLS LP 15 30.9738 0.000 A 9.16000e-03 2.50700e-05 ;phosphor, OPLS LOS 8 15.9994 0.000 A 2.56300e-03 1.86800e-06 ;ester oxygen, OPLS LP2 6 14.0270 0.000 A 5.87400e-03 2.26500e-05 ;RB CH2, Bergers LJ LP3 6 15.0350 0.000 A 8.77700e-03 3.38500e-05 ;RB CH3, Bergers LJ LC3 6 15.0350 0.000 A 9.35700e-03 3.60900e-05 ;CH3, OPLS LC2 6 14.0270 0.000 A 5.94700e-03 1.79000e-05 ;CH2, OPLS
В разделе [ nonbond_params ] Вы обнаружите такую строку - ;; parameters for lipid-GROMOS interactions. Удалите ее и все ниже расположенные строки из этого раздела. Так же Вы обнаружите взаимодействия, включающие в себя атом HW, так как все они являются нулевыми, то соответствующие строки можно либо удалить, либо заменить HW на H, что будет соответствовать правилам наименования GROMOS96 53A6.
Получившуюся папку Вы можете скопировать в директорию Gromacs, где расположены остальные поля, чтобы использовать модифицированное поле в будущем.
Минимизация энергии системы.
Cкачайте архив в интересующую Вас директорию.
wget http://molsim.org/sites/default/files/membraneDPPC.tar.gz
Архив содержит в себе файл топологии и три .mdp-файла.
Прежде, чем запустить минимизацию, необходимо создать .gro-файл.
editconf -f dppc128.pdb -o dppc128.gro
Начините минимизацию энергии:
grompp -f minim.mdp -c dppc128.gro -p topol_dppc.top -o em.tpr
И запустите молекулярную динамику:
mdrun -v -deffnm em
NVT и NPT ансамбли
Запуск молекулярной динамики в NVT и NPT ансамблях осуществляется аналогично запуску минимизации энергии.
Запустите сначала NVT.
grompp -f nvt.mdp -c em.gro -p topol_dppc.top -o nvt.tpr mdrun -v -deffnm nvt
А затем NPT.
grompp -f npt.mdp -c nvt.gro -p topol_dppc.top -o npt.tpr mdrun -v -deffnm npt

